NY STUDIE. En forskargrupp vid Göteborgs universitet har utvecklat en metod som gör det möjligt att se exakt var i genomet särskilda protein binder in. Metoden bygger på att proteiner kan påverka hur UV-ljus skadar DNA.
Metoden publiceras i tidskriften Nature Communications. Kerryn Elliott, medarbetare i Erik Larsson Lekholms grupp, är försteförfattare:
– Metod tillåter oss att förutsättningslöst kartlägga avtrycket av flera proteiner samtidigt. Den fångar interaktionen mellan proteiner och DNA i det ögonblick som UV-ljuset träffar det, säger Kerryn Elliott, som är forskare på Avdelningen för medicinsk kemi och cellbiologi vid Institutionen för biomedicin.
Tydliga inbindningsprofiler

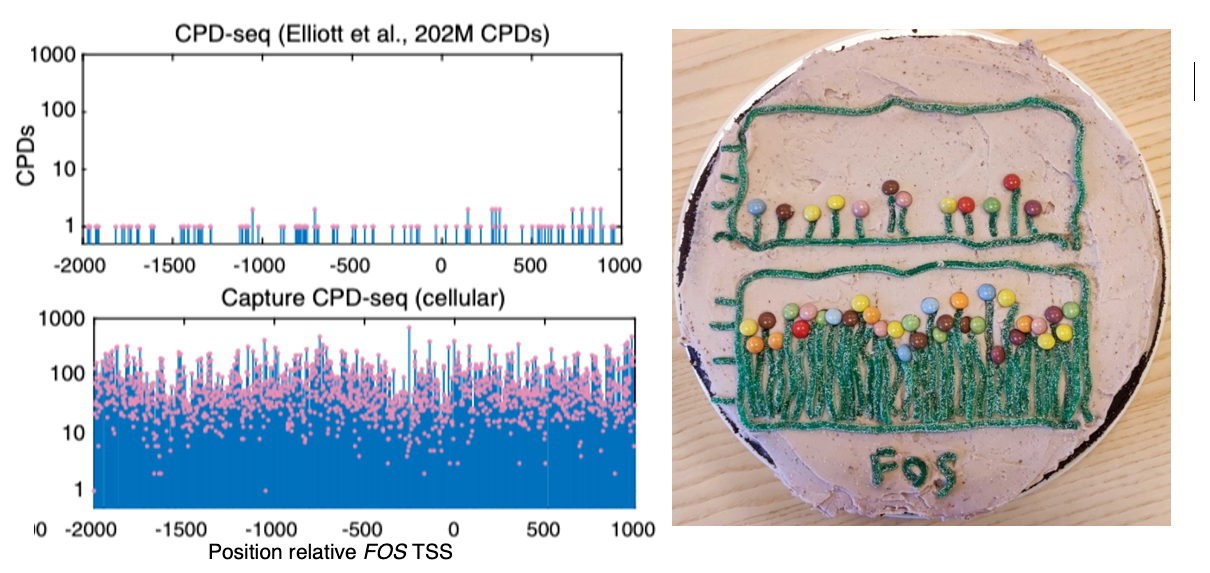
Gruppen utvecklade metoden delvis för att kunna studera varför mutationer ofta uppstår i promotorer i malignt melanom. Promotorer är sekvenser av baspar framför en gen som binder in olika proteiner för att reglera genens uttryck. Forskarna upptäckte att vissa proteiner har förmåga att kraftigt öka DNA-skadan från UV-ljus där de binder till genomet. Detta fenomen förklarar varför upprepade mutationer uppstår i promotorer i hudcancer, och talar emot att dessa bidragit till utvecklingen av cancersjukdomen. Metoden har för första gången möjliggjort noggranna mätningar av UV-skada i DNA på baspars-nivå, och kan också användas för att studera var i genomet som proteiner binder in.
– Vi får tydliga inbindningsprofiler för flera transkriptionsfaktorer. Vi kunde till och med träna en beräkningsmodell som kan förutsäga om en given sekvens är bunden eller inte baserat på dess signatur för UV-skada, vilket jag tycker är ganska coolt, säger Kerryn Elliott.
Parallellpublicering

Metoden, kallad Capture CPD-seq, innebär att en metod som kallas CPD-seq kombineras med en infångning (capture) av specifika promotorsekvenser, vilket möjliggör kvantitativa analyser av UV-skador för individuella baspar. Detta följs sedan av nästa generations sekvensering vid Clinical Genomics Gothenburg Core Facility. Den nya metoden innebär att mönster av UV-skador i DNA som binder till proteiner inuti celler kan jämföras med proteinfritt DNA. På så vis skapas ett slags fingeravtryck av UV-skador.
Förhoppningen är att metoden också ska kunna användas för att kartlägga olika DNA-strukturer och andra intressanta platser i genomet som har visat sig vara svåra att analysera i celler.
Först med att använda CPD-seq-metoden med UV-ljus var en forskargrupp vid Washington State University, som använde metoden för att studera UV-skador i jäst. Erik Larsson Lekholms grupp har anpassat denna för användning i mänskliga celler. Förra året insåg grupperna att de båda arbetade på liknande Capture CPD-seq-projekt, och de båda grupperna har därför skrivit två artiklar för Nature Communications, som publicerades samtidigt.
– Inom en snar framtid hoppas vi kunna använda den här principen för att profilera kompletta genom. Detta kan vara möjligtvis exempelvis i jäst, som har mycket mindre kromosomer än mänskliga celler, avslutar Kerryn Elliot.
Titel: Base-resolution UV footprinting by sequencing reveals distinctive damage signatures for DNA-binding proteins; Nature Communications, https://doi.org/10.1038/s41467-023-38266-2
AV: ELIN LINDSTRÖM












Grattis till så intressant arbete!